El listado, que solo contemplaba a Noruega, PaĂses Bajos, Suecia y Suiza, ahora tiene a Costa Rica como un actor clave en la ciencia global
En un momento crĂtico internacionalmente, una biotecnĂłloga y un microbiĂłlogo de la Facultad de MicrobiologĂa de la Universidad de Costa Rica (UCR) lograron lo inĂ©dito y colocaron el nombre de Costa Rica entre las potencias globales mĂĄs prestigiosas que estudian patĂłgenos âvirus y bacterias que pueden ocasionar la muerteâ, con el objetivo de generar, obtener y compartir informaciĂłn que contribuya a salvar vidas.
¿Sus nombres? El Dr. José Arturo Molina Mora y la Dra. Rebeca Campos Sånchez quienes, después de cinco meses de arduo trabajo, y con el apoyo de dos informåticos de la UCR, José Antonio Brenes Carranza y José Daniel Sånchez Castillo, posicionaron a Costa Rica como parte de la connotada plataforma web internacional Pathogens Portal.
Pathogens Portal es un espacio digital de elevado prestigio mundial del Pathogen Data Network âauspiciado por el Instituto Suizo de BioinformĂĄtica (SIB) y el Instituto Europeo de BioinformĂĄticaâ dedicado al acceso, al anĂĄlisis y a la distribuciĂłn de datos sobre agentes infecciosos de alto impacto.
Su objetivo es fungir como una gran base de datos colectiva cuya información puede ser usada para mejorar las herramientas diagnósticas, impulsar la generación de nuevos fårmacos que combatan eficazmente a los patógenos y potenciar terapias mås efectivas para los pacientes, especialmente, quienes han desarrollado resistencia a los tratamientos antibióticos y poseen pocas opciones terapéuticas disponibles.
âQue Costa Rica integre el Pathogens Portal es sumamente importante para el paĂs y un hito sin precedentes. En Costa Rica tenemos un desarrollo tecnolĂłgico muy eficiente, pero con un problema llamado la âmaldiciĂłn de la dimensionalidadâ. Esto significa que, aunque producimos datos masivos, no tenemos la misma capacidad de extraer la informaciĂłn relevante y los acumulamos con el anhelo de, algĂșn dĂa, analizarlosâ, comentĂł el Dr. Molina.
âComo consecuencia, hoy conocemos, por ejemplo, la existencia de 200 000 estructuras de proteĂnas validadas a nivel experimental. Pero, a nivel de secuenciaciĂłn genĂłmica, hay 250 millones. Esto significa que nos estamos perdiendo millones de oportunidades para entender los mecanismos biolĂłgicos de estos virus y bacterias a fin de descubrir nuevos fĂĄrmacosâ, aseverĂł el Dr. Molina.
Dr. JosĂ© Arturo Molina Mora, lĂder de la iniciativa desde la UCR
Hasta hace poco, Pathogens Portal solo contemplaba a las naciones de Noruega, PaĂses Bajos, Suecia y Suiza.
Gracias al trabajo visionario de ambos cientĂficos de la UCR, la red latinoamericana CABANAnet y del respaldo de la VicerrectorĂa de InvestigaciĂłn, Costa Rica hoy integra esa distinguida lista y es, oficialmente, reconocida como la Ășnica naciĂłn de CentroamĂ©rica en integrar el nodo regional del Pathogens Portal y, por el momento, el primero de LatinoamĂ©rica, pues luego se integrarĂĄn MĂ©xico y Chile. Estos nodos regionales tambiĂ©n son financiados por el SIB y el proyecto es coordinado por Aitana Neves.
âA travĂ©s del proyecto de CABANAnet vamos a fortalecer y ayudar a otros paĂses de la regiĂłn para que tambiĂ©n depositen datos en esta plataforma y, al mismo tiempo, vamos a favorecer las capacitaciones para que la gente conozca cĂłmo se pueden utilizar este tipo de herramientas. Para la UCR es fundamental ser un lĂder en este esfuerzo, porque sabemos que tendremos un gran impacto, no solo para nuestro paĂs, sino tambiĂ©n para toda la regiĂłn Latinoamericanaâ, apuntĂł la Dra. Campos, investigadora de la UCR y coordinadora de CABANAnet.
de un ecosistema cientĂfico colaborativo en AmĂ©rica Latina, impulsado por redes como CABANAnet.Â
Esta red ha demostrado cĂłmo la colaboraciĂłn regional puede transformar la investigaciĂłn en
salud,con mås de 60 intercambios académicos, 13 proyectos colaborativos y mås de 1 000 personas
capacitadas en anĂĄlisis genĂłmico, bioinformĂĄtica y ciencia abierta.
Dra. Rebeca Campos SĂĄnchez.
ÂĄUna esperanza!
El hecho de que Costa Rica integre el Pathogens Portal potenciarĂĄ una transformaciĂłn estratĂ©gica en el manejo y vigilancia de enfermedades infecciosas en el paĂs. Esto es esperanzador.
Actualmente, Latinoamérica estå entre las regiones con los niveles mås altos de bacterias resistentes, muy similar a la tendencia mundial.
Por ejemplo, la Ășltima RevisiĂłn de la Resistencia Antimicrobiana del Reino Unido indica que la resistencia de las bacterias estĂĄ generando el fallecimiento de casi 700 000 personas cada año en el mundo y, si la situaciĂłn no cambia, para el 2050 podrĂa cobrar la vida de mĂĄs de 10 millones en todo el orbe.
Esa proyecciĂłn se respaldĂł aĂșn mĂĄs a finales del 2022 con el Ășltimo informe emitido por la OrganizaciĂłn Mundial de la Salud (OMS). Este informe revelĂł altos niveles de resistencia en bacterias causantes de septicemias (infecciones potencialmente mortales), asĂ como una creciente resistencia de los patĂłgenos bacterianos mĂĄs comunes a varios de los tratamientos existentes.
Asimismo, en el 2024, comentĂł el Dr. Molina, se identificĂł que una de las bacterias infecciosas mĂĄs comunes en Costa Rica que genera diarreas y vĂłmitos, Escherichia coli (E. coli), ya estaba presentando resistencia a varios antibiĂłticos de uso frecuente y de Ășltima lĂnea de terapia.
En palabras mĂĄs sencillas, existe un alto riesgo de quedarnos sin fĂĄrmacos capaces de contrarrestar bacterias altamente dañinas para el ser humano, desde las mĂĄs comunes, como la E. coli, hasta las mĂĄs raras. Este panorama refuerza la urgencia de implementar, cuanto antes, estrategias de vigilancia genĂłmica y control antimicrobiano mĂĄs efectivas en el paĂs.
âUno de los principales aspectos que intentamos cubrir con este portal, y con nuestro equipo de trabajo en inteligencia artificial, genĂłmica y bioinformĂĄtica, es la fuga de datos. Sabemos que existen bacterias resistentes, pero esos genomas no se estĂĄn secuenciando y lo poco que se hace no se estĂĄ compartiendo para el uso y beneficio colectivoâ, resaltĂł el Dr. Molina.
¿Para qué sirven los anålisis genómicos en patógenos?
Los anĂĄlisis de genoma completo en el estudio de patĂłgenos (como bacterias o virus) sirven para conocer todo su ADN, es decir, su manual de instrucciones.
Båsicamente, es como leer un expediente que nos dice quién es el patógeno y qué puede hacer. Esta información nos ayuda a combatirlo mejor, elegir el tratamiento correcto, rastrear brotes y prevenir su expansión.
Al dĂa de hoy, agregĂł el experto, los paĂses de LatinoamĂ©rica individualmente no han aportado ni 100 genomas de cada tipo de bacteria crĂtica, lo cual es vital en las bases de datos mundiales para el desarrollo de nuevas terapias y diagnĂłsticos.
AsĂ, la incorporaciĂłn del paĂs en ese espacio apoyarĂĄ a las personas investigadoras, profesionales en salud y responsables de polĂticas pĂșblicas en el avance de iniciativas de salud, estudio y vigilancia de microorganismos patĂłgenos y de preparaciĂłn ante pandemias a travĂ©s del acceso equitativo y la colaboraciĂłn.
âMediante el portal podremos localizar patrones de comportamiento de los patĂłgenos a travĂ©s del tiempo y optimizar las estrategias diagnĂłsticas, especialmente, de aquellas bacterias resistentes para las cuales se agotan las opciones de tratamientoâ, reafirmĂł el experto.
âYa hay informaciĂłn accesible para todas las personas interesadas, como los 1 534 genomas depositados en las bases de datos del SARS-CoV-2, en el Pathogens Portal que estamos inaugurandoâ, añadiĂł la Dra. Campos.
âSi nosotros damos esta informaciĂłn en las bases de datos, los diseñadores de pruebas de anĂĄlisis a nivel global pueden incorporar nuestras secuencias y optimizar los proyectos a nivel de equipos automatizados que luego se traen a nuestras latitudes. Costa Rica estĂĄ siendo un hub para la regiĂłn, porque estamos trayendo las capacidades tecnolĂłgicas y de bioinformĂĄtica que antes no tenĂamosâ.Dr. JosĂ© Arturo Molina Mora.
Trabajo revolucionario
El posicionamiento de Costa Rica en Pathogens Portal es producto del apoyo de CABANAnet y del proyecto iPAT: Plataforma basada en GenĂłmica, BioinformĂĄtica e Inteligencia Artificial para el estudio de patĂłgenos aislados en entornos clĂnicos, de la UCR.
En los Ășltimos cinco años, la UCR ha sobresalido en el anĂĄlisis masivo de datos mediante el uso de la inteligencia artificial, lo cual ha sido determinante en el agrupamiento de virus y bacterias para definir grupos diversos y caracterizarlos. La IA tambiĂ©n optimiza la clasificaciĂłn de los patĂłgenos, lo cual tambiĂ©n agiliza la generaciĂłn de clases para su estudio.
âYa hemos hecho aplicaciones de IA en la UCR para el estudio de patĂłgenos. Uno es el caso del COVID-19. Hace tres años implementamos, en colaboraciĂłn con el Instituto Costarricense de InvestigaciĂłn y Enseñanza en NutriciĂłn y Salud (Inciensa), un estudio con datos de casi 19 000 pacientes en el cual utilizamos algoritmos de agrupamientoâ, comentĂł el Dr. Molina.
âAl concluir, logramos establecer siete perfiles clĂnicos que diferenciaban la sintomatologĂa y luego los comparamos con factores de riesgo, la carga viral diferida por la prueba molecular y, ademĂĄs, se comparĂł con las versiones del SARS-CoV-2, a fin ayudar a mejorar los abordajes terapĂ©uticos que daba el personal de saludâ, agregĂł el microbiĂłlogo.
Otras aplicaciones han sido con el perfil de citoquinas de pacientes con COVID-19. Las citoquinas son mensajeros que le dicen al cuerpo qué hacer para defenderse. En ese momento, la meta era entender mejor las diferencias en la recuperación de las y los pacientes.
Asimismo, y en colaboraciĂłn con el Massachusetts Institute of Technology (MIT), desde la UCR se efectuĂł una clasificaciĂłn para estudiar enfermedades crĂłnicas del tracto digestivo en un modelo de monos marmoseta, con importantes hallazgos que, en un futuro, podrĂa traducirse en mejores terapias.

Gracias a estos esfuerzos, CABANAnet ha generado datos valiosos sobre microbiomas, resistencia antimicrobiana y patĂłgenos emergentes que ahora, mediante el Pathogens Portal, se podrĂĄn conectar con iniciativas globales. Esto fortalece la vigilancia genĂłmica regional y mejora la capacidad de respuesta ante amenazas sanitarias.
Talento informĂĄtico
Aunque el diseño original del Pathogens Portal surgió en Europa, el nodo costarricense fue cuidadosamente tropicalizado para responder a las realidades locales.
A diferencia de los modelos de Chile o MĂ©xico, el equipo informĂĄtico de la UCR, compuesto por JosĂ© Antonio Brenes Carranza y JosĂ© Daniel SĂĄnchez Castillo, adaptĂł la estructura base del nodo suizo a las necesidades de investigaciĂłn y gestiĂłn de datos propias del paĂs.
Por eso, se desarrollaron herramientas internas especĂficas que permiten la colaboraciĂłn directa entre investigadores locales y la integraciĂłn eficiente de datos ya existentes, como los provenientes de proyectos impulsados por CABANAnet.
âSiempre tenemos esa necesidad de contar con portales que nos permitan divulgar la informaciĂłn. En el caso de Costa Rica, ya tenemos diferentes proyectos e investigaciones que se han desarrollado. Por lo tanto, ya se contaba con esa informaciĂłn y era simplemente la necesidad de tener el portal como tal con algunos aspectos particularesâ, declarĂł JosĂ© Antonio Brenes.
Justamente, uno de esos aspectos particulares del nodo de Costa Rica es que no solo ofreciera una interfaz funcional y eficiente, sino que incluyera mecanismos de gestiĂłn que permitieran a diversos actores cientĂficos subir y administrar contenido especializado.
Para lograrlo, la plataforma fue diseñada con un enfoque modular, lo que permite su futura expansión con nuevas visualizaciones, dashboards interactivos y conexiones con bases de datos nacionales e internacionales.
AdemĂĄs, se contemplan futuras integraciones de analĂtica avanzada que permitirĂĄn mostrar datos procesados directamente desde las investigaciones costarricenses.
âEn esta primera etapa sacamos una primera versiĂłn estable que cumpliera con la funcionalidad. Para la segunda etapa esperamos que la plataforma se siga alimentando y siga aumentando los nĂșmeros a nivel de bases de datos y a nivel de visitas, que vamos a empezar a monitorear para ver quiĂ©nes nos visitan y cuĂĄl es el impacto de esta plataformaâ, complementĂł JosĂ© Daniel SĂĄnchez.
El equipo tĂ©cnico trabajĂł contra el reloj para que Costa Rica fuera el primer paĂs Latinoamericano en lanzar su nodo del Pathogens Portal. El desarrollo se completĂł en menos de seis meses, desde el primer trimestre de 2025, con una primera versiĂłn estable que cumple con los requisitos mĂnimos para su funcionamiento.
âNos apresuramos bastante porque ya habĂa otras plataformas avanzadas y querĂamos ser los primeros en LatinoamĂ©rica en salir al aireâ, señalĂł JosĂ© Antonio Brenes, quien destacĂł el apoyo constante de la Dra. Campos y el Dr. Molina para lograr este hito.
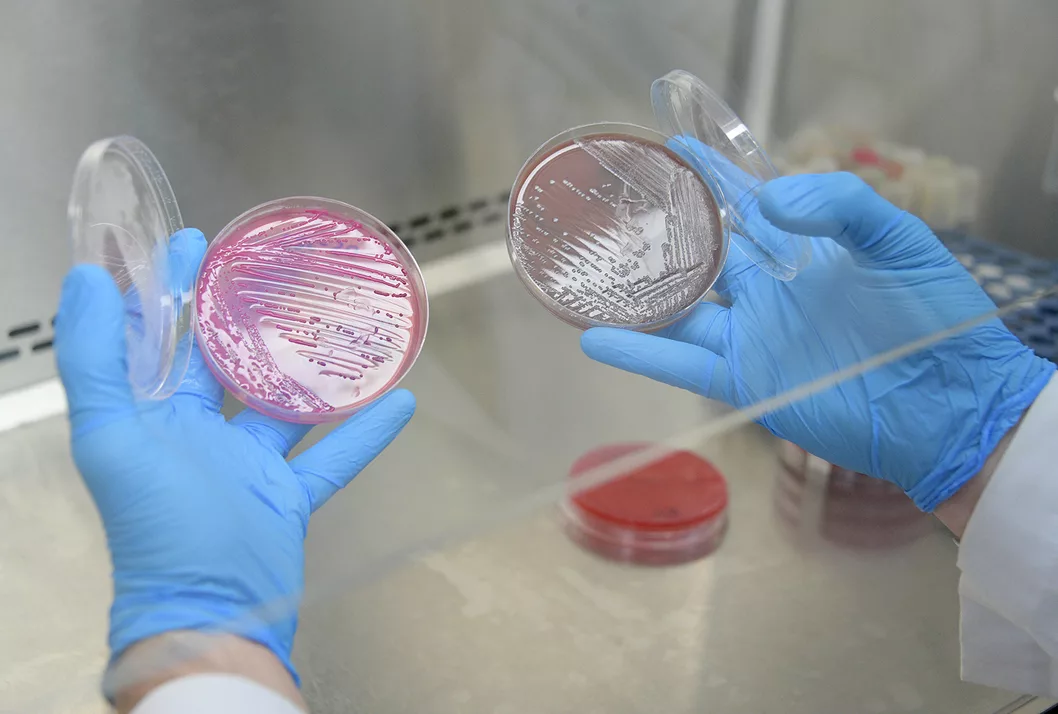
La resistencia bacteriana a los antibiĂłticos es una amenaza creciente que dificulta el tratamiento de infecciones comunes, poniendo en riesgo la salud global y aumentando la necesidad de nuevos enfoques en la lucha contra las bacterias. En la imagen, placas de Petri con cultivos bacterianos, una herramienta para estudiar y comprender el fenĂłmeno.
¿Qué sigue?
Con el proyecto iPAT de la UCR y el Pathogens Portal, el énfasis estarå en la resistencia a los antimicrobianos.
Si bien todos los patĂłgenos son importantes, el Dr. Molina aclarĂł que existe una clasificaciĂłn de prioridades que incluye a un grupo de bacterias resistentes a los carbapenĂ©micos: los fĂĄrmacos antibiĂłticos mĂĄs avanzados y de Ășltimo recurso para tratar infecciones graves en pacientes hospitalizados.
âLa idea con el portal es integrar el trabajo y estudios de diferentes laboratorios, propiamente a nivel clĂnico relacionado con salud, trabajar en redes y adoptar una perspectiva de una salud con mejores herramientas para el diagnĂłstico y de seguimiento en diferentes ambientesâ, expresĂł el Dr. Molina.
Si bien la incorporación al portal es reciente, desde la UCR ya se brindaron mejoras diagnósticas a la Caja Costarricense de Seguro Social (CCSS) para identificar pacientes resistentes a los antibióticos y adaptar las terapias con el objetivo de aumentar su eficacia.
âLos hospitales pĂșblicos nos reportaron una inconsistencia de los resultados para la detecciĂłn de enzimas que estudian carbapenĂ©micos. Estas pruebas se le hacen al paciente a fin de administrar el tratamiento antibiĂłtico mĂĄs adecuado y eficaz. Resulta que la prueba molecular de PCR no detectaba un gen en particular y la prueba fenotĂpica sĂ daba positiva. Esto se ha reportado en MĂ©xico, Puerto Rico, Brasil, entonces no Ă©ramos los Ășnicos con este problemaâ, dijo el Dr. Molina.
âAl secuenciar, nos dimos cuenta de que el gen sĂ estaba presente, pero diferente, lo que nos da un falso negativo en el PCR. Con esa primera aproximaciĂłn, hicimos luego 119 aislamientos y propusimos mejoras en el diagnĂłstico que ya tiene la CCSS y que ya estĂĄn siendo de mucha ayuda para la vida de las y los pacientesâ, expuso el Dr. Molina.
Por el momento, y desde el proyecto iPAT, la UCR estå trabajando con diez instituciones aliadas por tres años, a fin de extender la representación de Costa Rica en bases de datos, optimizar las pruebas diagnósticas, agilizar los anålisis de genoma completo y proveer información a nivel de uso de herramientas de inteligencia artificial.
El siguiente paso es que Costa Rica sea un centro lĂder en AmĂ©rica Latina que fortalezca la seguridad sanitaria global mediante la promociĂłn de la investigaciĂłn innovadora sobre patĂłgenos, el fomento de la cooperaciĂłn internacional y la garantĂa de un reparto equitativo de beneficios e informaciĂłn para prevenir y responder eficazmente a las pandemias.
En cuanto a la parte informåtica, las próximas fases incluirån mejoras en visualización de datos, interacción con los usuarios y mayor interoperabilidad con otras plataformas de ciencia abierta.
âQueremos que no sea solo un repositorio de datos, sino una herramienta de anĂĄlisis e interpretaciĂłn Ăștil para la comunidad cientĂfica nacional e internacionalâ, concluyeron los desarrolladores.
ÂĄGrandes avances UCR!
En el marco del Proyecto iPAT, las y los investigadores costarricenses han logrado optimizar al mĂĄximo los recursos y han generado resultados de alto impacto. A continuaciĂłn, algunos de los logros mĂĄs relevantes:
1. AnĂĄlisis genĂłmico masivo optimizado
Se logrĂł establecer un nĂșmero mĂnimo de secuencias para analizar el genoma bacteriano, con una eficiencia equivalente a haber generado mil veces mĂĄs secuencias, lo que genera un ahorro en costos de secuenciaciĂłn.
2. IdentificaciĂłn de grupos especĂficos de alto riesgo
Se detectaron clones del patĂłgeno Acinetobacter baumannii en el Hospital San Juan de Dios, asociados a brotes clĂnicos y se identificaron genes relacionados con mecanismos de resistencia relevantes en contextos hospitalarios para ayudar a las y los pacientes.
3. ComparaciĂłn genĂłmica internacional
Se realizó una comparación exhaustiva entre cepas locales y clones internacionales de la bacteria A. baumannii, lo que reveló similitudes con linajes de alta capacidad de diseminación. Esto posiciona a Costa Rica en un contexto global de vigilancia genómica, permitiendo anticipar riesgos epidémicos.
4. Uso de inteligencia artificial
A partir del genoma bacteriano de la bacteria Pseudomonas aeruginosa, se logrĂł inferir el fenotipo de resistencia antimicrobiana mediante modelos de IA, alcanzando un desempeño de hasta un 80âŻ% de precisiĂłn. Asimismo, se impulsan modelo especĂficos con una exactitud de 76-80âŻ% para predecir resistencia a carbapenĂ©micos en una bacteria crĂtica. Estos modelos no sustituyen las pruebas fenotĂpicas tradicionales, pero son una herramienta poderosa para el anĂĄlisis masivo y rĂĄpido de datos genĂłmicos.
5. Perturboma
Se explorĂł el concepto de âPerturbomaâ, es decir, la respuesta molecular de un organismo ante condiciones de estrĂ©s, con implicaciones directas para comprender adaptaciones a antibiĂłticos y ambientes hostiles. Esta âinteligencia internaâ de los microorganismos es clave para desarrollar estrategias terapĂ©uticas mĂĄs efectivas.
6. Plataforma PABS (PatĂłgenos, AnĂĄlisis, BioinformĂĄtica y SecuenciaciĂłn)
Se avanza en la implementaciĂłn de la plataforma PABS, diseñada para integrar los datos de genĂłmica, bioinformĂĄtica e inteligencia artificial. Su uso permitirĂĄ consolidar una base de datos nacional robusta y facilitar el acceso a informaciĂłn crĂtica para la toma de decisiones clĂnicas y de salud pĂșblica.
Esta noticia fue escrita por Jenniffer Jiménez Córdoba
Periodista de la Oficina de ComunicaciĂłn Institucional de la Universidad de Costa Rica.
Publicada originalmente en UCR | Noticias.
(Reproducida con fines informativos y educativos.)


